 |  |
伯豪生物官網(wǎng) www.ukdirect.cn 新官網(wǎng)消息 單細胞 RNA 測序科研服務來了
SMART-seq

2012 年,由美國和瑞典的科學家共同開發(fā)了稱為 Smart-Seq(Switching mechanism at 5’end of the RNA transcript)的技術 [1]。在 2013 年,Nature Methods 雜志上報告了新的方案,被稱為 Smart-Seq2[2],隨后在 2014 年他們有公布了詳細的操作流程 [3]。獲取單細胞并裂解細胞后,含有 oligo-dT 的引物與 mRNA 的 poly- A 結(jié)合,逆轉(zhuǎn)錄酶 MMLV 進行逆轉(zhuǎn)錄,逆轉(zhuǎn)錄酶到達 mRNA 5’末端時,MMLV 的末端轉(zhuǎn)移酶活性會在一鏈 cDNA 的 3'末端增加額外的胞嘧啶,這時 TSO 引物 3' 末端的 rGrG+ G 結(jié)合到首鏈末端的胞嘧啶,然后以首鏈 cDNA 為模版進行延伸合成互補的第二鏈,全長 cDNA 經(jīng)過 PCR 擴增后進一步進行測序文庫的構(gòu)建。

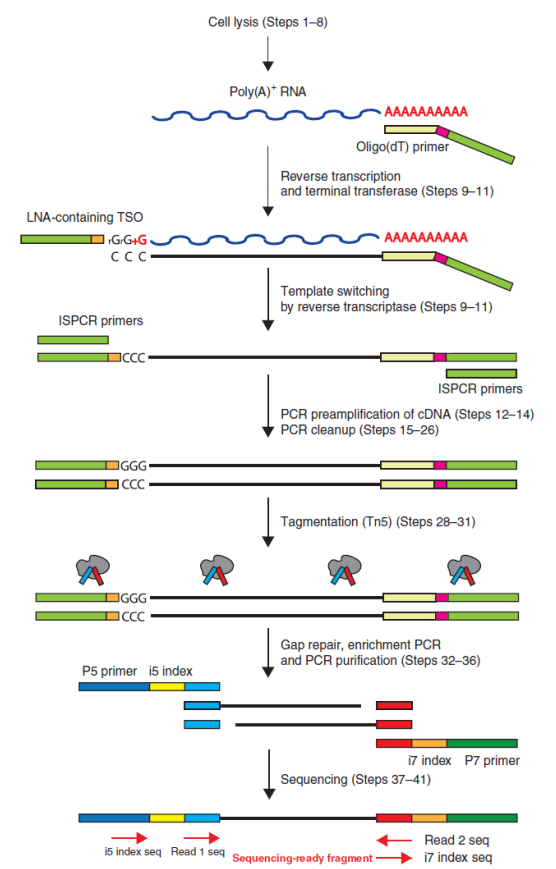
▲SMART-seq2 技術原理
伯豪生物提供:單細胞轉(zhuǎn)錄組測序、生信分析整體科研技術服務,伯豪生物單細胞轉(zhuǎn)錄組測序優(yōu)研服務商,24 小時咨詢服務熱線:021-58955370

1、起始量低:1 個細胞起始,適用于難以滿足 10X Genomics 等平臺起始細胞量要求的樣本。
2、覆蓋度高: 測序覆蓋 cDNA 的全長序列,每個細胞能夠獲得上萬個基因的表達。
3、信息個性化: 除了獲得基因表達信息,數(shù)據(jù)能夠用于可變剪接,cSNP 等分析,以及帶 poly- A 結(jié)構(gòu)的 lncRNA 研究。
伯豪生物提供:單細胞轉(zhuǎn)錄組測序、生信分析整體科研技術服務,伯豪生物單細胞轉(zhuǎn)錄組測序科研服務商,24 小時咨詢服務熱線:021-58955370

文庫結(jié)構(gòu)

圖 SMART-seq2 文庫示意圖
伯豪生物提供:單細胞轉(zhuǎn)錄組測序、生信分析整體科研技術服務,伯豪生物單細胞轉(zhuǎn)錄組測序科研服務商,24 小時咨詢服務熱線:021-58955370
建議測序深度及參數(shù)
指標 | 參數(shù) |
測序深度 | 6Gb/cell |
測序類型 | PE150 |
伯豪生物提供:單細胞轉(zhuǎn)錄組測序、生信分析整體科研技術服務,伯豪生物單細胞轉(zhuǎn)錄組測序科研服務商,24 小時咨詢服務熱線:021-58955370

1、類型:新鮮組織,原代細胞,細胞系等。
2、來源:血液提取、磁珠富集、流式富集、組織解離等。
3、樣本量:單個細胞(或微量細胞)。
4、保存運輸:細胞放入 SMART-seq 試劑盒指定的裂解液中,-80°C 保存,干冰運輸。

| 序號 | 文件類型 | 查閱 |
| 1 | 【畫冊】單細胞全長轉(zhuǎn)錄本測序解決方案 | 點擊查看 |
| 2 | 【畫冊】石蠟樣本(FFPE)單細胞轉(zhuǎn)錄組測序解決方案 | 點擊查看 |
| 3 | 【畫冊】單細胞測序解決方案 | 點擊查看 |

